SARS-CoV-2
El ISCIII detecta la circulación de 800 linajes de Ómicron, la variante del SARS-CoV-2 dominante en España
El texto se ha copiado correctamente en el portapapeles
El Instituto de Salud Carlos III (ISCIII), como coordinador de la Red Nacional de Laboratorios Españoles de Secuenciación Genómica de SARS-CoV-2 (Relecov), detectó cerca de 800 linajes y sublinajes de Ómicron, la variante del virus de la covid-19 predominante en España desde 2022, según un informe dado a conocer este lunes en el que se analizó la evolución de la circulación de este virus entre los meses de octubre de 2022 y octubre de 2023.
La Red Relecov está formada por 49 laboratorios distribuidos por todas las comunidades autónomas, coordinados por el Centro Nacional de Microbiología del ISCIII, y que realiza desde 2021 la secuenciación genómica, el estudio y la vigilancia del SARS-CoV-2 en España.
De esta forma se obtiene un conocimiento clave para seguir el comportamiento y evolución del coronavirus, apoyar su estudio epidemiológico y, entre otras cuestiones, dado que el virus evoluciona y se adapta, optimizar el desarrollo de vacunas.
Durante el año de estudio de este informe se caracterizaron más de 47.000 virus SARS-CoV-2, lo que permitió conocer su genoma y realizar el seguimiento de la circulación de la variante Ómicron y sus diferentes linajes y sublinajes, que fueron surgiendo como consecuencia de los cambios genéticos que continuamente experimenta el virus.
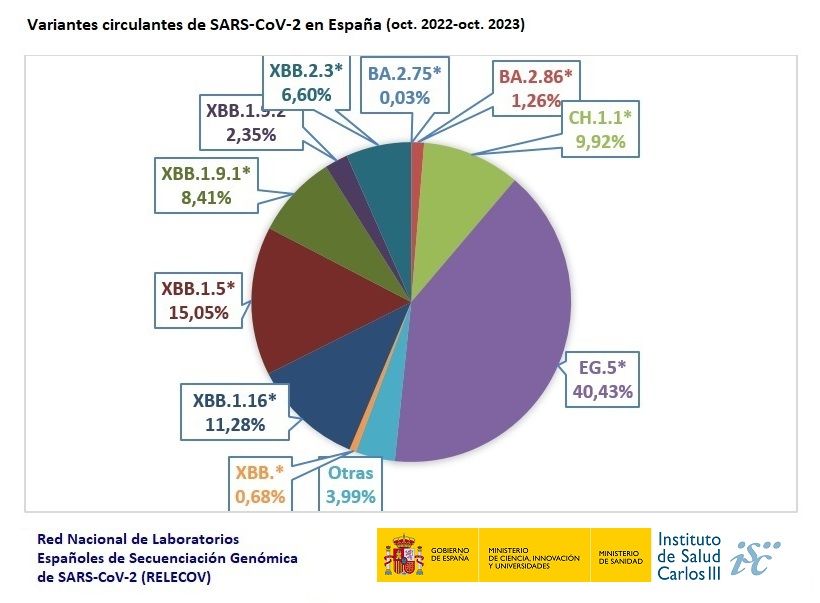
Se dio a conocer la circulación de cerca de 800 linajes y sublinajes diferentes de Ómicron en España, asignados a diferentes clados del SARS-CoV-2. Los clados son grupos filogenéticos que definen la evolución biológica del virus y su comportamiento, algo fundamental para la vigilancia y su aplicación en salud pública.
SECUENCIACIÓN Y VIGILANCIA
Todos los linajes y sublinajes detectados fueron descendientes de la variante Ómicron, predominante en España a partir de 2022. El informe reveló datos de secuenciación genómica, vigilancia de variantes, análisis de mutaciones y estudios filogenéticos.
En el periodo estudiado, siguiendo las directrices establecidas por la Organización Mundial de la Salud (OMS), las variantes circulantes de interés (VOI) detectadas comprendieron los linajes XBB.1.5* , XBB.1.16* y EG.5*. Por su parte, las variantes bajo monitorización (VUM) comprendieron los linajes correspondientes a BA.2.75*, CH.1.1*, XBB*, XBB.1.9.1*, XBB.1.9.2*, XBB.2.3* y BA.2.86*.
En cuanto a la evolución de las variantes, se observó una disminución muy significativa en la circulación de diferentes variantes, entre ellas BA.2.75*, XBB.1.5*, XBB.1.9.1* y XBB.1.9.2*, y un aumento en la circulación de otras, como es el caso de EG.5*, CH.1.16* y CH.1.1*.
(SERVIMEDIA)
22 Ene 2024
ABG/gja



