Investigación
Descubren un mecanismo molecular asociado al desarrollo de la esclerosis lateral amiotrófica
El texto se ha copiado correctamente en el portapapeles
Investigadores del Consejo Superior de Investigaciones Científicas (Csic) descubren un mecanismo proteico relacionado con el desarrollo de la esclerosis lateral amiotrófica (ELA) y la demencia frontotemporal, entre otras patologías neurodegenerativas.
Así se desprende de los resultados del estudio, publicados en la revista ‘Nature Communications’, que “podrían en un futuro ayudar en el diagnóstico médico de estas enfermedades”, según los autores de la investigación.
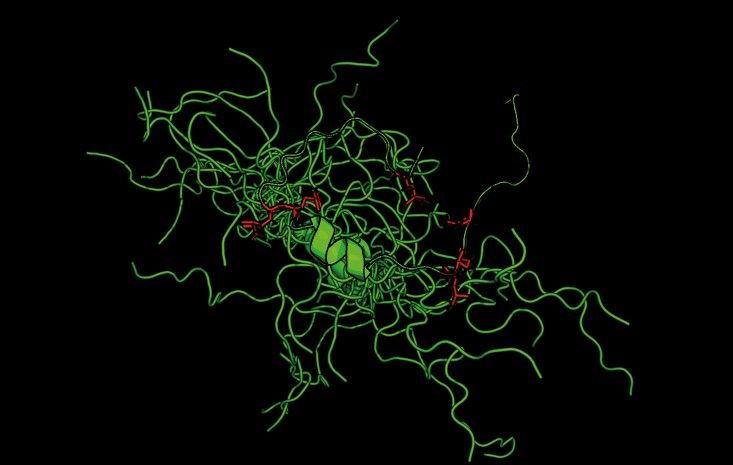
“Hemos demostrado que unas nuevas estructuras en un prionoide (proteína que adopta una estructura infectiva que se autorreplica pero que, a diferencia de un prión, no es transmisible), inducidas por modificaciones químicas irreversibles que ocurren en condiciones de estrés, alteran fuertemente los mecanismos de la proteína y su relevancia en el desarrollo de enfermedades”, explicó el investigador del Csic Javier Oroz, del Instituto de Química Física Rocasolano.
Añadió que “en concreto, la proteína TDP-43 al modificarse, presenta una vía molecular diferente a la que se pensaba hasta ahora que era el origen de su toxicidad en la esclerosis lateral amiotrófica”.
Estas nuevas estructuras, mucho más dinámicas y desordenadas, escapan del control de las chaperonas moleculares, impiden la formación de condensados por separación de fases y forman unos agregados con una morfología significativamente distinta.
Según este especialista, “dado que esa modificación, entre otras, ocurre en el desarrollo de la enfermedad, y que su efecto estructural es muy severo, demostramos que es necesario tenerla en consideración si se realizan estudios de proteínas para entender los mecanismos moleculares de las enfermedades”.
“Además, el efecto tan pronunciado de las modificaciones en la agregación de TDP-43 abre la puerta al estudio sistemático de distintas variantes de TDP-43 como reactivos de diagnóstico para la esclerosis lateral amiotrófica”, subrayó Oroz.
Los resultados de este estudio tienen implicaciones también en el campo de la biología estructural. "Dado que la estructura de las proteínas cambia drásticamente al sufrir una modificación, estas estructuras no pueden predecirse con programas de inteligencia artificial de uso habitual en el estudio del plegamiento de proteínas como AlphaFold", destacaron los autores de la investigación.
En este trabajo también participaron especialistas del Centro de Biología Molecular Severo Ochoa (centro mixto del Csic y la Universidad Autónoma de Madrid) y de la Universidad de Hong Kong. Esta investigación ha sido posible gracias al uso de los espectrómetros de Resonancia Magnética Nuclear del Laboratorio Manuel Rico (Instituto de Química Física Rocasolano), una Infraestructura Científica y Técnica Singular (ICTS) del Csic.
(SERVIMEDIA)
01 Mar 2023
CAG/gja



